感染症の数理シミュレーション(1)からの続き
動機
政府は,NHKテレビを通じて「ここ1,2週間が瀬戸際」で「感染のピークシフトを」と訴えている。ところが,具体策としては,多少具合が悪くても自宅で寝ておけというもので,それ以上の(中国のような)大胆な政策出動には及び腰だ。北海道や和歌山県などでは感染者が報告されてきたが,大阪や京都で感染者がほとんどいないのはどうみても不自然だ。SNSのうわさでは,保健所や病院でたらい回しにされながら検査を拒否されているという。ごく一部の例で感染拡大をアピールしながら,厚生労働省の検査基準と各地方自治体の保健所の連係プレーで,医療資源保護や東京五輪のために検出の押さえ込みを図っていると見えてしまう。
ところで,日本の医療資源の限界とはどの程度のもので,感染によってこれがどの程度枯渇することが想定されているのか,あるいは,ピークシフトの時間スケールはどうなのかという半定量的な情報はほとんど出されていない。重症化した感染者が病院で病床について治療されることを考えてみる。
厚生労働省の医療施設動態調査(平成30年2月)によれば,一般診療所を除く日本全国の病院の療養病床を除く病床数は約123万床なので国民100人に1床の割合だ。なお
月末病床利用率というのも厚生労働省から報告されており,全国平均で約80%である。したがって,コロナウィルスの対応可能病床数は100人に0.2床ということになる。実際にはすべての病床が対応可能ではないから,100人に0.1床(1万人に10人)を上回る重症者が発生すれば危険というオーダーか。
そこで,今回の新型コロナウイルス感染症の特徴を満たすSEIRをもとにした数理モデルを作成して,重症者や死亡者のシミュレーションを行ってみる。なお,
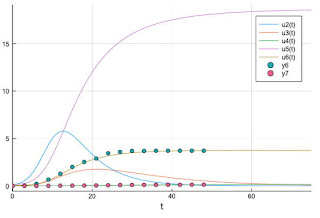
PFNの丸山宏さんが,
林祐輔さんが公開したSEIRモデルを参考に,
ダイアモンド・プリンセスにおけるCOVID-19発症日別報告数を観測データとして,
最適化ツールOptunaを用いてパラメターフィッティングを試みている。丸山さんは,SIRモデルを仮定し(感染しない潜伏期間がないもの),N=3711人の集団に対し,初期の感染者(発症していない人を含む)I0,感染率β(Nで割っていない),感染者がある日に発症する率α,発症期間γをパラメータ推定した。発症率が4%で,初期感染者が209名,一人当たり感染数(Nβ)は41だ(βの定義に注意)。
モデル
SEIRモデルを少し修正する。免疫を持たないものが感染する部分は同じである。この軽症感染者は一定期間をへて,回復者又は重症感染者にいたる。重症感染者は,一定期間をへて回復者または死亡者にいたる。重症感染者は病院または自宅で隔離されるためそれ以上の感染源とはならない。死亡者も同様だ。軽症感染者は無症状かもしくは軽い風邪程度の症状なので普通に活動し,免疫非保持者を感染させる可能性がある。この重症感染者の数を病床数と比較することで,医療資源の破綻の程度が判断できる。
図 SEIRモデルを修正したSIIDRモデルの概念図
$\dfrac{du_1}{dt} = -\beta \dfrac{ u_1*u_2}{n}$
$\dfrac{du_2}{dt} = \beta \dfrac{ u_1*u_2}{n} -\dfrac{u_2}{\alpha_1} -\dfrac{u_2}{\alpha_2}$
$\dfrac{du_3}{dt} = \dfrac{u_2}{\alpha_2} -\dfrac{u_3}{\gamma_1} -\dfrac{u_3}{\gamma_2}$
$\dfrac{du_4}{dt} = \dfrac{u_3}{\gamma_2}$
$\dfrac{du_5}{dt} = \dfrac{u_2}{\alpha_1} +\dfrac{u_3}{\gamma_1}$
計算
$n$=10,000人の集団を考える。免疫非保持者$u_1$が9,999人,軽症感染者$u_2$が1人の初期状態から約1年間の推移をシミュレートする。軽症感染者の感染期間を14日として,その96%が回復し,4%が重症化すると仮定する。すなわち,$\alpha_1=14/0.96$ ,$\alpha_2=14/0.04$。さらに,重症感染者の感染期間を7日として,その90%が回復し,10%が死亡するとする。感染者の死亡率は0.4%になる。すなわち,$\gamma_1=7/0.90$ ,$\gamma_2=7/0.10$ 。感染率$\beta$をパラメタとして,0.1から0.3まで変化させた。ここで得られる結果$u_1, \cdots u_5$ はすべて集団1万人あたりの人数になる。そこで,重症感染者数 $u_3$と1万人当たりの受け入れ可能病床数(10床)を比較して,医療資源の枯渇の様子を調べる。
- - - - - - - - - - - - - - - - - - - - - - - - - - - - - -
using DifferentialEquations
using ParameterizedFunctions
using Plots; gr()
sky = @ode_def SIIDR_model begin
du1 = -β*u1*u2/n # u1:Noimmunity(Susceptible)
du2 = β*u1*u2/n -u2/α1 -u2/α2 # u2:Mild(Infected-a)
du3 = u2/α2 -u3/γ1 -u3/γ2 # u3:Serious(Infected-b)
du4 = u3/γ2 # u4:Dead
du5 = u2/α1 +u3/γ1 # u5:Recovered
end n α1 α2 β γ1 γ2
n=10000.0 #total number of population
α1=14.0/0.97 #latent to recovery (days/%)
α2=14.0/0.03 #latent to onset (days/%)
β=1.0 #infection rate
γ1=7.0/0.90 #onset to recovery (days/%)
γ2=7.0/0.10 #onset to death (days/%)
function epidm(n,α1,α2,β,γ1,γ2)
u0 = [9999.0,1.0,0.0,0.0,0.0] #initial values
p = (n,α1,α2,β,γ1,γ2) #parameters
tspan = (0.0,360.0) #time span in days
prob = ODEProblem(sky,u0,tspan,p)
sol = solve(prob)
return sol
end
β=0.3
@time so=epidm(n,α1,α2,β,γ1,γ2)
println(β," ",so[4,45]," ",so[3,19])
plot(so,vars=(0,3))
β=0.2
@time so=epidm(n,α1,α2,β,γ1,γ2)
println(β," ",so[4,40]," ",so[3,19])
plot!(so,vars=(0,3))
β=0.15
@time so=epidm(n,α1,α2,β,γ1,γ2)
println(β," ",so[4,34]," ",so[3,20])
plot!(so,vars=(0,3))
β=0.12
@time so=epidm(n,α1,α2,β,γ1,γ2)
println(β," ",so[4,30]," ",so[3,22])
plot!(so,vars=(0,3))
β=0.1
@time so=epidm(n,α1,α2,β,γ1,γ2)
println(β," ",so[4,28]," ",so[3,24])
plot!(so,vars=(0,3))
- - - - - - - - - - - - - - - - - - - - - - - - - - - - - -
結果
上記モデルを用いたピークシフトのふるまいは,次のグラフのような結果となる。
図:1万人当たりの重症感染者数の感染率β依存性,左からβ=0.30, 0.20, 0.15, 0.12, 0.10
重症感染者数$u_3$のグラフから得られた結果と死亡者数$u_4$の値をまとめると次のようになる。
表:感染率βと1万人当たりの重症感染者のピーク及び1年後の1万人当たり死亡者数
β ピーク日 ピーク病床
死亡者
0.30
50
54
30
0.20
80
38
28
0.15
125
23
25
0.12
185
13
20
0.10
280
7
13
重症感染者のふるまいの感染率$\beta$への依存性は非常に大きい。$\beta$が0.12を越えると,必要なピーク病床数は簡単に受け入れ可能病床数の10を超えてしまい,医療パニックが発生する。これが,感染開始1ヶ月から半年の間に起こる(当然オリンピックの時期を含む)。非常に激しければ短期(3ヶ月)で収束するが混乱は限度を超える。感染率を押さえこんだとしても影響が長期化することは避けられない。
付記
肺炎による死亡は,悪性新生物や心疾患についで日本人の死因の第3位になっており,年々死亡率(10万人当たり死者数)が上昇している。1万人当りに換算すると,年に10人が肺炎で死亡していることになる。上記のβ=0.10の場合の13人はこれと同じオーダーであることから,もし中間的な過程が隠蔽されたとしても,1年後には明らかに新型コロナウィルス肺炎による肺炎死亡者の急増が観測されるはずである。
感染症の数理シミュレーション(3)に続く